ゲノムの特定の配列(プロモーター)を認識して結合するタンパク質群がある。これらのタンパク質は時に大きな複合体を形成する。
たとえばTATA配列に結合するTBPを中心としたタンパク質複合体である。
これらはRNApolymeraseとさらに結合する。
しばしば分子生物学の教科書では、とても静的な漫画でこの状態が描かれている。まず
TBPがやってきて結合して、次に○○がやってきて結合して、そして二重鎖が乖離して....というように。
しかし実際にはここではDNAもそれらタンパクも激しくブラウン運動をしているはずで、くっついたり離れたりの平衡状態にあるはずだ(近年の一分子を観る技術は、こうした実際を見せてくれるようになってきている!)。
この平衡状態(のもつ平均的な様子)は次のような化学平衡で表すことができる。
RNApolymerase + promoter ↔ complex
これらの量的な関係は
Eq. 3
Kp
gene, cell = [complex
gene, cell]/[RNApolymerase]/[promoter
gene, cell]
ただしKpは平衡定数(promoterのK)、[complexgene, cell]はそのプロモーターでの複合体の濃度(または形成頻度と考えるべきかも)、[RNApolymerase]は核内で自由にしている酵素の濃度(たぶん一定だろう)、[promotergene, cell]は結合していないプロモーターの濃度。
この式はさらに次のように近似できる。
Eq. 4
[complex
gene, cell] ≈ a
gene, cell×P
0×Kp
gene, cell[RNApolymerase]
ただしaはその領域でのDNAの活性を表すファクター。(
epigeneticな調節の項を参照)。近似は[complex]/[promoter]がゼロに近いだろうことに由来。この仮定は
Kimura et. al. 1999
などの測定からの推定。
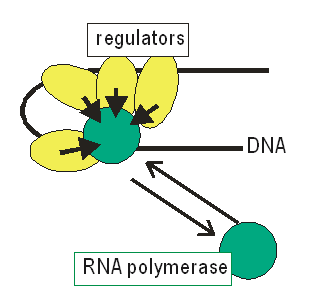
Kpは、原核細胞であるなら、プロモーター部位の塩基配列でかなりの部分が決定される(DNAとRNApolymeraseが直接に会合するからだ)。真核細胞では、RNApolymeraseはTBPをはじめとしたタンパク性の因子ごしにpromoterと会合する。
こうやってDNAに(やや不安定ながら)結合しているタンパク質群が、その場所とRNApolymeraseとの親和性を決定する。これらとRNApolymeraseの会合はもっとずっと弱い力でおきているらしい。というのも、核内のRNApolymeraseのほとんどは、何かに結合するのではなくて単独で存在しているからだ(Kimura et. al. 1999
)。もし強い力で結合するのなら、むしろそれらタンパクとRNApolymerase はもっと大きなコンプレックスを形成してDNAとの結合部位を探すようなことになるだろう(それは事実とは異なるようだ)。
こうしたDNAと、DNA結合性タンパク質の平衡定数は少しずつ測定されるようになってきていて、
九州工業大学の皿井先生がデータベース化されている。
http://gibk26.bse.kyutech.ac.jp/jouhou/3dinsight/recognition.html
このデータべースは統合データベースプロジェクトのポータルからもたどることができる。
http://lifesciencedb.jp/
DNAに結合したそれらタンパクはRNApolymeraseとTBPとの会合に影響する。あるものはよりこの会合を安定させるだろうし、あるものはむしろ阻害するだろう。それはつまり、何がどこに(どんな向きで)結合するのかによる現象であろう。この平衡状態を決定するのはとりもなおさず(TBPを含む)DNAタンパク質複合体である。それらの個々の、polymeraseと接する(あるいは電気的に影響する)成分の、その影響の一つ一つを、RNApolymeraseが会合することによって放出される自由エネルギー変化ΔGのひとつの成分ΔΔGとして表すことができる。
ここでは単純にするために、因子Aと因子BがそれぞれRNApolymerase に与える影響を独立したものと考えている。もしかしたら因子Aは因子Bと隣り合うと立体構造が変化して、別の働きを持つことになるのかもしれないけれど、現在のところそうした事例は知られていないと思う(のでモデルを不必要に複雑化させていない)。
ここでこのΔΔGを決めているのは、だから、以下の2つである。
ひとつは、何がそこにある(ない)のかである。
因子Aが核内に[因子A]なるモル濃度で存在するとき、その因子はどのくらいの確率でそのプロモーターに結合しているか?
これは時間的な確率としても、あるいは細胞の集団で構成される組織に関して、ある瞬間にどのくらいの割合の細胞でそれが結合しているのかとしても考えられる。つまり、結合している・していないことそのものは二値的だけど、
トランスクリプトームとして考えるときはこれは連続する分布をもつ値である。
Eq. 5
[cis-regulator
cis, gene, cell] ≈ a
gene, cell×P
0×Kc
cis, gene[regulator
cis]
cell
[cis-regulator]は、ある遺伝子のある塩基配列に、タンパク性因子が結合している状態を濃度で表した値。[regulatorcis]はそのcis因子に結合するタンパク性因子の核内の活性濃度。近似は[complex]/[regulator]がゼロに近いだろうことに由来---はるかに多い分子がブラウン運動しながら核内にある、これは一分子観察で観察される。Kcは平衡定数。
もうひとつは、その因子Aの形状(そして荷電)と、プロモーター内の位置である。
これらはつまり、RNApolymeraseと「何がどういった位置で」関係するのかを決定する。基本的にひとつの生物において、因子の性質はあまり変化しないだろう。またもちろんプロモーター内の配列は不変である。そこで、この性質(因子の形状と因子の結合位置)は不変であろう。
これをkr(regulator のk)という比例定数で表すことにする。
Eq. 6
ΔΔGp
cis, gene, cell = Kr
cis, gene ×[cis-regulator
cis, gene, cell]
これで、ある遺伝子のある配列に結合する、ひとつのregulatorの、RNApolymeraseに対する働きが記述できたことになる。
もちろんこうしたcis因子が遺伝子ごとに複数存在するはずである。それぞれの因子の働きが独立である、ないし、それらの働きを因子ごとに分解して考えることができると考えると(後述)、
それらの因子の働きは結局、これら自由エネルギーの総和として表される。Eq.5と6からΔGp (promoter のΔG)を求めると、
Eq. 7
ΔGp
gene, cell = Σ a
gene, cell×P
0×kr
cis, gene×Kc
cis, gene[regulator
cis]
cell
このGibbs 自由エネルギー変化量をつかって、Eq. 3の平衡定数を求めることができる。すなわち、
Eq. 8
Kp
gene, cell = exp(-
ΔGp
gene, cell/R/T)
これとEq.4を使うと、プロモーターとRNApolymeraseとの会合を、ΔGpの関数として表すことができる。
Eq. 9
[complex
gene, cell] ≈ a
gene, cell P
0 [RNApolymerase]×
exp(-
ΔGp
gene, cell/R/T)
aはその領域のDNAの活性定数、P0は核内のDNA濃度、[RNApolymerase]は核内でフリーなポリメラーゼ濃度、Rは気体定数、Tは絶対温度。これらは定数か、あまり変化しない性質を持つと考えられる。そこで変化しうるのはΔGpだけである。enhanceosomeと呼ばれるような巨大なコンプレックスは、おそらくとても低い値を持つのだろう。
