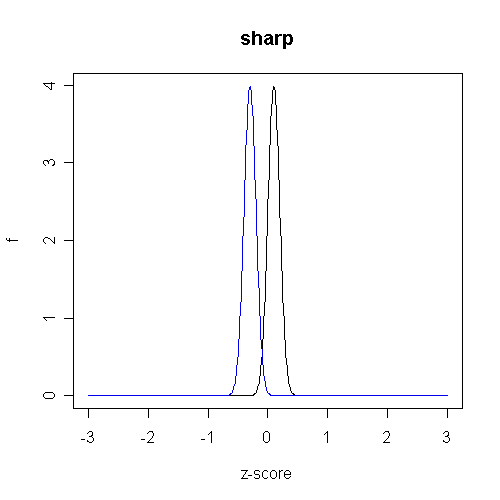
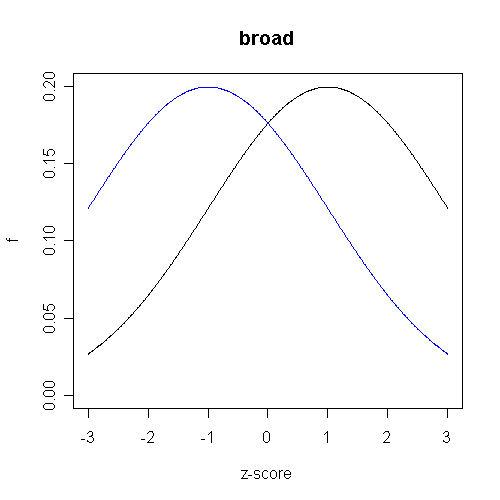
マイクロアレイは、分子生物学の測定方法のなかでは際立って項目数(スポット数)が多い。
そのため、偽陽性False Positiveの数も多くなる。これは困ったことだ。
偽陽性を根絶することは不可能だけど、
自分たちが耐えられるだけの数にコントロールすることはできる。
これはパラメトリック統計の得意分野のひとつである。
ここではそのための方法を概説する。
マイクロアレイで偽陽性が増えるのは、項目数が多いからである。
もし組織Aと組織Bの間に差があるかどうかが問題になっていて、
それを2組のマイクロアレイデータから検定しようとしているのなら、
これは(スポット数ぶんの)多重検定である。
けれど、これは設問がそもそもナンセンスだ。
どのトランスクリプトームも、少しずつ違っているものだから。
調べている組織が増えていって、たとえば組織Aから組織Gまでの間で
一度でも変動したことがある遺伝子を見つける、
なんていう際には多重検定の問題が生じる。
まあしかしこれはよくあるケースであって、マイクロアレイが特別なわけではない。
(そして、この種の試みをする場合には、偽陽性は深刻な問題にならないことが多い。
これはいわゆる宝探しであって、有望な候補は必ず追試されることになる)。
それは何か別の数学的なフレームワークを必要とするだろう。
帰無仮説が棄却できないときは黙るしかない。
差はなかったのだとは言えない。